TEST PRENATALE NON INVASIVO. DNA FETALE IN SANGUE MATERNO
NON SOLO TRISOMIA 21
TUTTO IL GENOMA
CONVALIDATO IL TEST CHE STUDIA TUTTI I CROMOSOMI
NIPT-DNA-CARIOTIPO
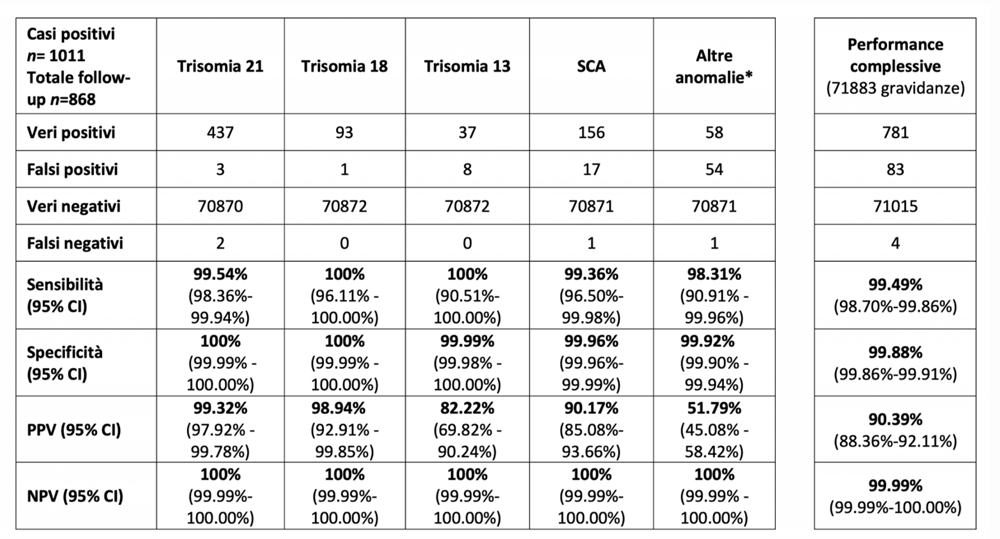
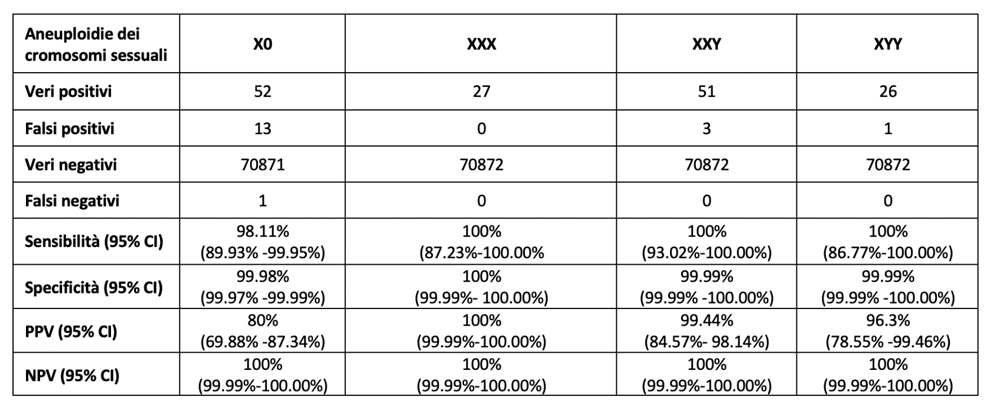
Il test sul DNA fetale che analizza ora tutto il cariotipo molecolare. Dalla trisomia 21 con 1,4:1000 casi a tutte le anomalie (aneuploidie) con 4:1000 casi.
Il Test Prenatale Non Invasivo NIPT che indaga il DNA fetale presente nel sangue materno a partire dalla 10 settimana di gestazione migliora le proprie capacità passando dalla analisi della Trisomia 21 fetale (e Trisomie 13 e 18 già individuabili ecograficamente) alla analisi di tutto il cariotipo.
La stessa tecnica genetica che si usa con prelievo di villi coriali o liquido amniotico (villocentesi o amniocentesi). La differenza fra CARIOTIPO-NIPT-DNA e cariotipo da amniocentesi/villocentesi sta nella accuratezza, simabile in 99,8/100.
La causa dei falsi positivi e negativi è prevalentemente dovuta alla discordanza feto placentare, dato che il DNA fetale nel sangue materno origina dalla placenta.
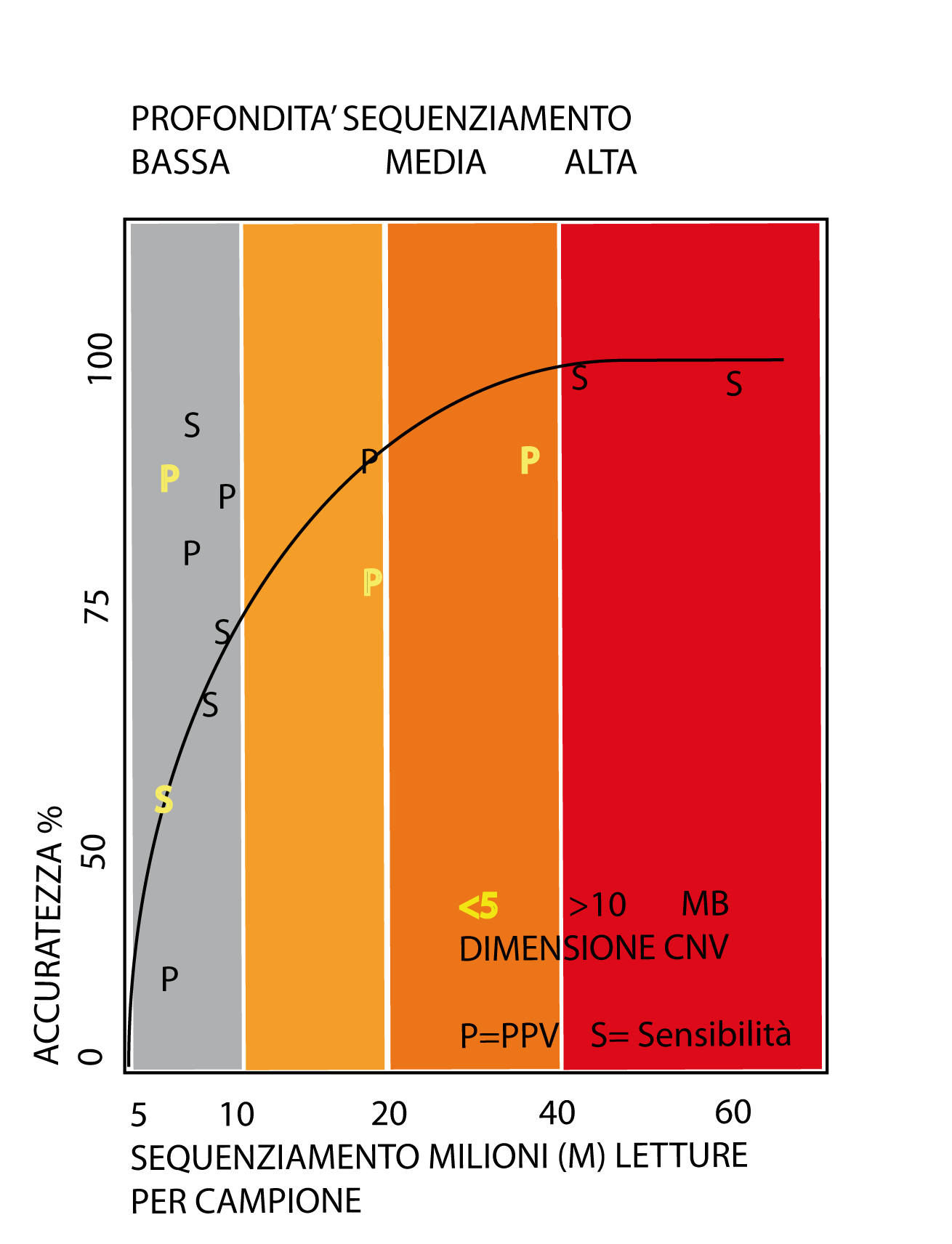
Tuttavia la sensibilità (capacità di individuare anomalie) e la specificità (veri positivi sul totale) come pure il PPV (Valore Predittivo Positivo, cioè i veri positivi su tutti i positivi) variano con diversi fattori: dimensione della sequenza di DNA coinvolto, profondità delle letture (coverage), frazione fetale, algoritmo con z-score dedicato.
Una sintesi, dedotta dalle principali recenti elaborazione può essere rappresentata:
Reference1, Reference2, Reference3, Reference4, Reference5.
A partire dal primo trimestre di gravidanza è presente nel circolo ematico materno DNA libero di origine fetale (cffDNA, cell free fetal DNA), che può essere prelevato in maniera non-invasiva ed utilizzato per lo studio di alcune patologie fetali. Il DNA libero presente nel circolo materno origina dalla lisi di cellule materne e cellule placentari. A partire dalla V settimana di amenorrea il citotrofoblasto placentare si ancora alla decidua parietale uterina; le arterie spirali deciduali irrorano le lacune presenti tra la decidua e la placenta; il citotrofoblasto invade e tappezza le pareti delle arterie spirali e le rimodella. Il ricambio delle cellule del trofoblasto che tappezzano le pareti delle arterie spirali, mediato dalle citochine, libera il DNA. I frammenti di DNA fetale degradato hanno dimensioni di circa 180 bp (paia di basi) e sono sospesi nel plasma arterioso. Sebbene il cffDNA del trofoblasto sia già isolabile alla V settimana, solo a partire dalla X settimana la sua quantità è sufficiente per indagini diagnostiche. La sua percentuale varia tra 0 e 25%, con una media, a 12 settimane, del 12%. Anche l’apoptosi degli epiteli materni libera frammenti di DNA nel plasma che concorrono ad una commistione di DNA materno e feto-placentare. Il cffDNA scompare dalla circolo materno poche ore dal parto, probabilmente attraverso l’escrezione renale. Si stima che i campioni ematici materni contengano, dopo la X settimana, circa 65.000 frammenti di 180 paia di basi del DNA del cromosoma 21. YMD Lo nel 1997 descrisse per primo la presenza del cromosoma Y nel plasma di alcune madri incinte di feti di sesso maschile, utilizzando l’analisi del cfDNA (cell free DNA).Le tecniche impiegate nell’analisi del cfDNA plasmatico si basano sulla digital PCR, Massively parallel shotgun sequencing (MPSS) dell’intero genoma o di sequenze bersaglio, SNP-based NGS, Digital analisys of selected regions (DANSR). L’analisi può perciò sequenziare i frammenti dell’intero genoma o selezionare solo i frammenti di alcuni cromosomi di interesse. La linea di sequenziamento (sequencing lane) è l’unità operativa del test; ogni linea può leggere 100 Gbasi di DNA (100 milioni di reads). L'applicazione di algoritmi dedicati permette di analizzare i frammenti di cfDNA ed è stata mirata alla previsione del rischio delle più comuni aneuploidie (cromosomi 21, 13 e 18 e cromosomi sessuali). Le tecniche attuali analizzano i frammenti cfDNA senza differenziare i frammenti di origine materna da quelli di origine fetale, in modo da consentire di disporre di quantità consistenti di DNA. Questi test sono definiti NIPT (Non Invasive Prenatal Test) e sono considerati “non diagnostici” proprio perché basati su una commistione di DNA di diversa origine, materna e placentare.La specificità dei test su cffDNA, negli studi che hanno arruolato oltre 10.000 campioni, rivela una percentuale di falsi-positivi <1/1.000 e conferma la percentuale di discordanze feto-placentari emersa dalle analisi cromosomiche effettuate sul citotrofoblasto. La percentuale di falsi-negativi nella popolazione afferente al test è <1/100-1.000 (sensibilità 99%) e conferma la percentuale di discordanze feto-placentari emersa dalle analisi cromosomiche sul citotrofoblasto. Nel 2015 il Ministero della Salute ha emesso le Linee Guida (download il documento nella barra a sinistra) che definiscono la validazione degli obbiettivi dell'indagine NIPT, i percorsi di scelta e prescrizione, nonche i modelli di consenso alla analisi da sottoporre alla donna.
La grande mole di dati raccolti con il diffondersi dei test ha consentito di migliorare le valutazioni di sensibilità e specificità, particolarmente distinguendo la affidabilità analitica (tecniche NGS e algoritmi) da quella biologica nel confronto trofoblasto vs. feto.
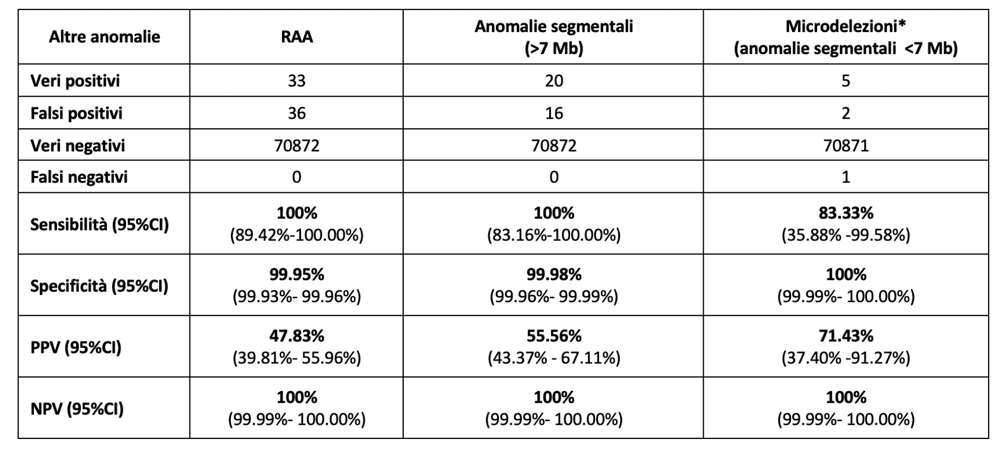
Il miglioramento delle tecniche di sequenziamento e il raffinamento degli algoritmi ha recentemente permesso di ridurre tempi e modalità di preparazione dei campioni di DNA (eliminazione PCR, automazione, double end sequencing, raffinamento dei fattori di normalizzazione). Conseguenza è stata anche la possibilità, applicando il Whole Genome Sequencing, di ricercare le anomalie cromosomiche più fini, duplicazioni e delezioni, specialmente di dimensioni superiori a 10Mb. La ricerca di CNV è stata presentata in alcuni studi retrospettivi e in almeno una review prospettica con risultati di rilievo. Nel 2019 la ditta Illumina ha validato e messo in commercio il kit tecnico per la esecuzione della analisi di sequenza genomica, che consente la esecuzione del cariuotipo molecolare.Riportiamo qui la sintesi attuale dello stato dell'arte con alcune tabelle messe a punto dallo staff Plurigentest.